ILMSImage Attribute Calculation
Índice |
 ILMSImage Attribute Calculation
ILMSImage Attribute Calculation
Introdução
O ILMSImage Attribute Calculation faz parte do ILMSImage plug-in para QuantumGIS e é utilizado para a derivação e cálculo de atributos baseados em células e para a integração das camadas de dados adicionais.
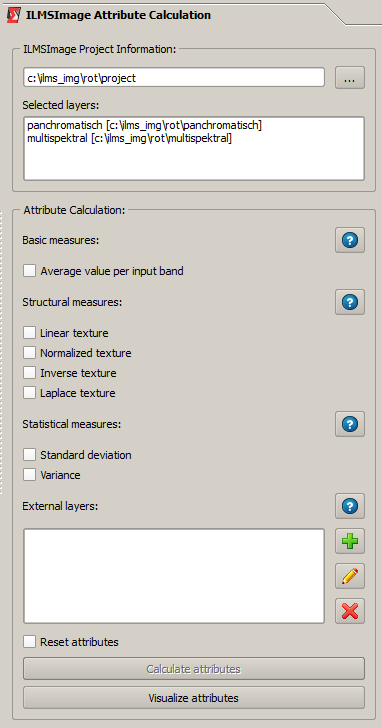
Semelhante a vários painéis ILMSImage que consistem de dois componentes,ILMSImage Project Information na parte superior e as ferramentas propriamente ditas na parte inferior.
Cálculo de Atributos
Informações Preliminares
...
Sumário
O painel possibilita as seguintes funções para o usuário:
- Cálculo do atributos padrão baseados em células: Além dos recursos de atributos básicos de geometria celular, diferentes características estruturais e medidas estatísticas fazem parte dos parâmetros implementados. Após o cálculo, esses são resumidos na forma de uma tabela de atributos.
- Adicionando layers de dados adicionais: As camadas de dados correspondentes são selecionadas pelo usuário, um identificador é atribuído e adicionado às camadas de dados existentes. Se necessário, o tipo de dados é adaptado automaticamente e os dados selecionados adicionais são cortados em termos de espaço.
- Visualização de atributos selecionados: Para este efeito, os atributos que são resumidos em uma tabela de banco de dados são transformados em dados raster e preparados no QuantumGIS para a visualização.
Cálculo de Atributos baseados em células
A fim de obter-se atributos específicos para as amostras a partir da combinação da geometria celular e dos dados de entrada selecionados, os parâmetros relevantes têm de ser ativados clicando-se na caixa de seleção correspondente. Os atributos padrão são valores básicos, características estruturais, parâmetros estatísticos e o implicitamente calculado características da geometria celular.
| Tipo de Atributo | Atributo | Nome GUI | Designação Interna | Descrição |
|---|---|---|---|---|
| Base | Valor médio por banda | Average value per input band | <layer_id> [_ <numerador>] |
Para cada faixa de entrada um valor médio é calculado a partir de todos os pixels da imagem resumida em uma célula. A designação interna corresponde a um identificador que é selecionado para a banda correspondente (no exemplo: pancromática ), no caso de dados multi-canal de entrada esses são completados por um número de canal (no exemplo: multi-spectral_1 , multi-spectral_2 , etc.)
|
| Estrutura | Textura Linear | Linear texture | linear |
A ativação desta medida cria a textura de primeiro grau em um Kernel 3x3. O algoritmo calcula a textura para cada célula individualmente e ignora todos os pixels da imagem que estão fora da célula atual. Como todas as características estruturais também a textura linear usa o primeiro canal que é exibido nas informações do projeto como base para o cálculo. |
| Textura normalizada | Normalized texture | normalized |
A textura normalizada usa o mesmo algoritmo que a textura linear, mas normaliza o resultado com o valor médio de todos os pixels da imagem da célula atual. Como todas as características estruturais também a textura normalizada usa o primeiro canal que é exibido nas informações do projeto como base para o cálculo. | |
| Textura Inversa | Inverse Texture | inverse |
Ao ativar a textura inversa o Inverse Difference Moment (Haralick et al. (1973), S. 619) é calculado no nível de pixel. A textura inversa usa o mesmo algoritmo que a textura linear e a normalizada, mas, finalmente, calcula o valor inverso do diferenças quadradas. Como todas as características estruturais também a textura inversa usa o primeiro canal que é exibido nas informações do projeto como base para o cálculo. | |
| Textura Laplace | Laplace texture | laplace |
Esta característica estrutural usa o kernel do Laplace modificado para o realce de contraste para células individuais. O parâmetro aumenta o resultado com um kernel genérico que reage a máximas de contraste local com distância espacial pequena (2-5 pixels). Como resultado, as linhas isoladas são reduzidas e regiões são preencidas com contrastes espacialmente densos. | |
| Estatísticas | Desvio padrão | Standard deviation | stddev |
Ao ativar esta opção é calculado o desvio padrão de todos os pixels reunidos em uma célula do primeiro canal exibido nas informações do projeto. |
| Variância | variance | variance |
Ao ativar esta opção, é calculado a variância de todos os pixels reunidos em uma célula do primeiro canal exibido nas informações do projeto. | |
| Geometria celular | Tamanho celular | Sem ligação GUI | size |
O tamanho de uma célula em pixels do canal de dados de entrada com maior definição. |
| Perímetro Celular | perimeter |
O perímetro de uma célula em pixels do canal de dados de entrada com maior definição. | ||
| Comprimento Celular | length |
O comprimento de uma célula em pixels do canal de dados de entrada com maior definição. | ||
| Largura da célula | width |
A largura de uma célula em pixels do canal de dados de entrada com maior definição. |
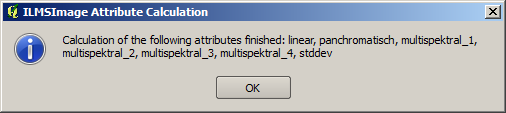
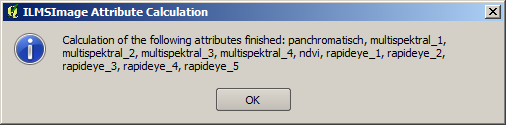
Após os atributos selecionados terem sido calculados, eles são apresentados resumidamente em uma tabela e ficam disponíveis para análise adicional utilizando ILMSImage. O usuário recebe um aviso sobre o fim do processo e um resumo dos atributos calculados.
Exportar e Visualizar Atributos baseada em Células
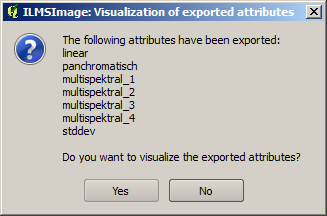
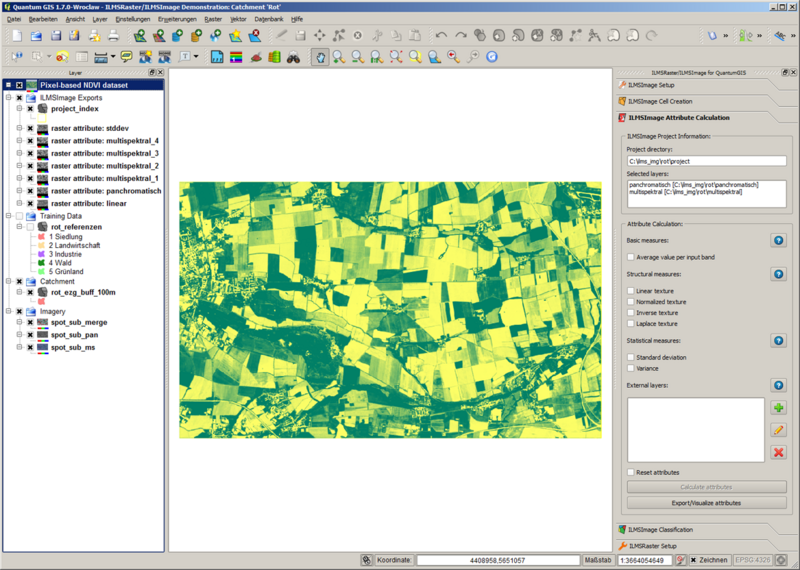
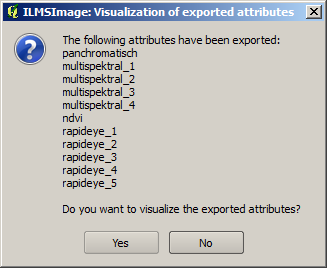
Para o controle visual, o plug-in possibilita exportar os atributos selecionados como camadas raster depois de terem sido calculados e mostrá-los posteriormente no atual Projeto QuantumGIS. Conseqüentemente, os atributos necessários são selecionados e o processo é iniciado, clicando-se em Export/ visualize attributes. Em seguida, o ILMSImage cria conjuntos de dados raster dos atributos e armazena-os no exports- diretório do projeto ILMSImage atual. A conclusão do processo é indicado para o usuário em uma janela de mensagem.
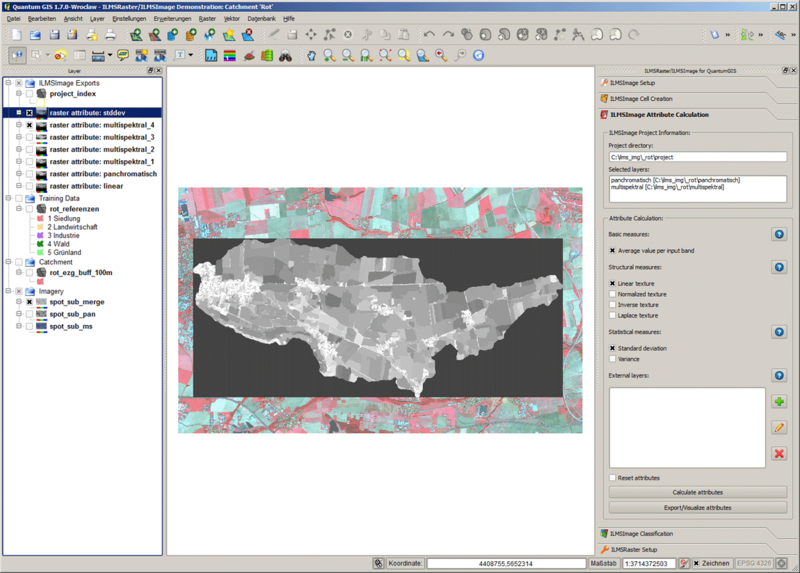
A janela também oferece a possibilidade de visualizar todos os atributos exportados automaticamente. Para este efeito, os conjuntos de dados raster são carregados no projeto QuantumGIS atual, transferidos para o grupo-chave para ILMSImage Exports e sua visualização é adaptada através de uma Standard Deviation Stretch. O resultado é representado em uma exibição de mapa como esta:
Se a solicitação correspondente é negada, os atributos exportados podem ser adicionados ao projeto QuantumGIS atual individual e separadamente pelo usuário. Neste caso, a visualização precisa ser modificada pelo usuário também.
Trabalhando com Camadas de dados externas
Adicionando e Editando camadas adicionais de Dados
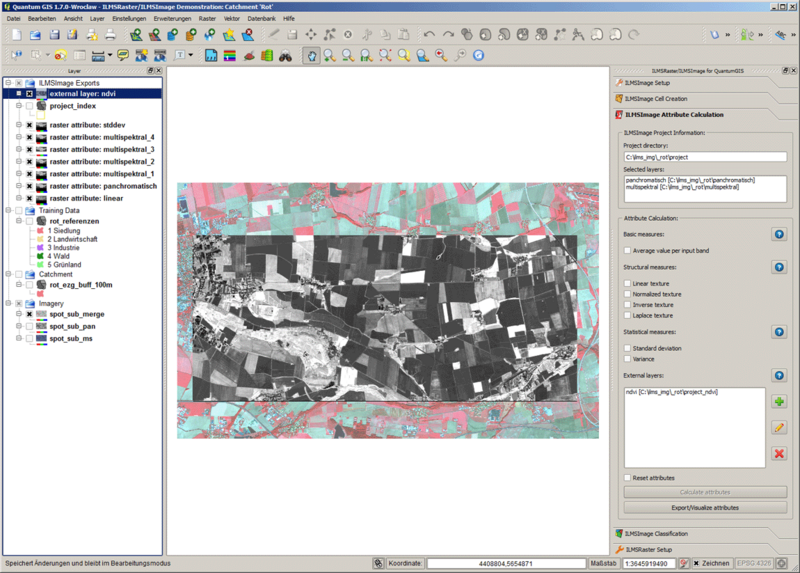
Além dos atributos padrão mencionados, as camadas de dados externas podem ser integradas no processo de cálculo de atributos. Para a integração basicamente todo conjunto de dados disponíveis em forma de raster é adequado. Na integração, dentro de cada camada e para cada célula, um valor médio é calculado e finalmente adicionado à tabela de atributos. Isto significa que atributos adicionais correspondem a o acima descrito atributo básico - no que diz respeito a ordenança de cálculo – a única diferença é que as camadas de dados adicionais não influenciam a criação de células, já que estas estão concluídas. Esta função é, portanto, adequada para a integração de níveis adicionais multi-espectrais (ou combinações destes), que são necessários como atributos para um análise adicional, mas não para uma derivação das áreas de análise, ou seja células, para o processo de trabalho. Um exemplo simples é o Índice de Diferença de Vegetação Normalizada (Normalized Vegetation Difference Index, NDVI), que é uma medida para a atividade da vegetação. Ele pode ser calculado através de dois canais que estão no campo visual e no campo próximo infravermelho do espectro eletromagnético. Como camada de dados adicionais de um NDVI baseado em pixel está disponível. A imagem seguinte mostra áreas de alta atividade de vegetação nas cores verde e áreas de baixa atividade em tons de amarelo.
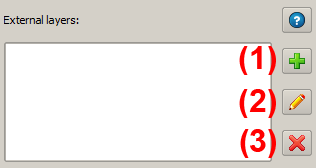
Este conjunto de dados pode agora ser definido como uma camada de dados externos - para tal, ele não precisa estar carregado no QuantumGIS, basta somente conhecer o caminho correspondente. Para a definição é usado o campo correspondente do painel. A ferramenta permite definir novas camadas externas (1), editar camadas existentes (2) e excluir as camadas externas de dados do projeto (3).
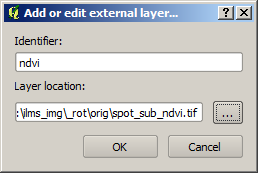
Para criar uma nova camada (layer) externa, uma caixa de diálogo aparece após clicar-se no ícone correspondente. Torna-se possível selecionar a fonte de dados correspondente no sistema de arquivos e atribuir um identificador. Note-se que um identificador único que não for selecionado já está sendo usado para outras camadas de dados ou para um dos atributos padrão (veja tabela acima). Para o conjunto de dados NDVI de exemplo ficaria assim:
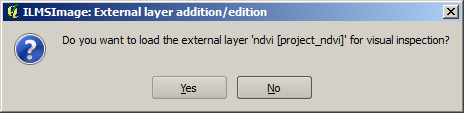
Após a confirmação, o conjunto de dados é copiado para o diretório do projeto para permitir que o ILMSImage tenha acesso exclusivo aos arquivos de origem para a criação de células de maneira análoga. Se necessário, ele é então ajustado para a área de teste. Para possiblitar o controle visual, o ILMSImage oferece a opção de carregar os resultados desses processos em um mapa:
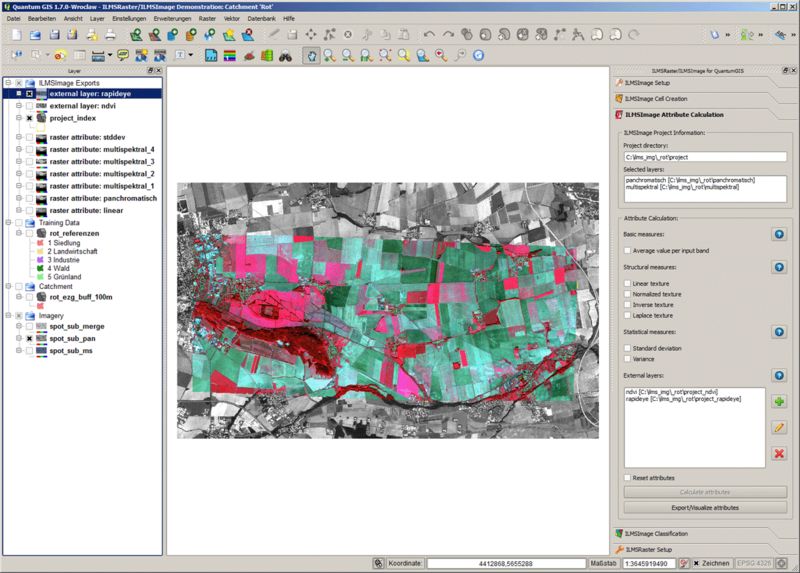
Depois de uma resposta afirmativa da pergunta, o conjunto de dados externo é visualizado através de um estiramento Standard Deviation Stretch; a gama de cores para os dados originais não é aplicada. Uma vez que essa representação só é usada para o controle visual, o conjunto de dados correspondente é excluído da visualização do mapa antes da execução de um cálculo de atributo. Além disso, o nível adicional gerado aparece na lista correspondente do painel de ferramentas. Na imagem a seguir uma área de alta atividade de vegetação é mostrada em cores claras, áreas de baixa atividade são representados em cores escuras:
Um conjunto de dados que já foi estabelecido pode ser editado clicando-se no ícone correspondente. É possível alterar o identificador e modificar o caminho do arquivo de origem.
Escala automática do tipo de dados de camadas de dados externas
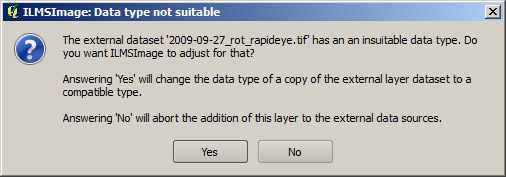
O ILMSImage só permite dados de entrada que estão disponíveis no domínio byte (8bit, valores de cinza entre 0 e 255). Se uma camada não-compatível é escolhida durante a definição de um nível de dados externos, o ILMSImage pergunta se essa camada deve ser ajustada automaticamente ao domínio aceitável. Se, por exemplo, uma parte de uma cena 16bit RapidEye deve ser integrada, a seguinte mensagem aparece :
Quando o pedido estiver confirmado, o conjunto de dados é escalado no domínio 8bit e é carregado – opcionalmente – para a visualização do mapa para controle visual.
Cálculo de Atributos Usando Camadas Externas de Dados
Os conjuntos de dados definidos externos estão disponíveis para o cálculo de atributos como a fonte de dados original e por isso são tratados como camadas equivalentes. Isto é válido para o cálculo real e a visualização das camadas calculadas.
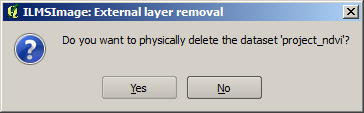
Excluindo Camadas Dados Externos
Para remover uma camada de dados externa a partir do projeto, deve se selecioná-la da lista de todas as camadas externas e clicar no ícone apropriado no painel. O ILMSImage então pergunta se o conjunto de dados relacionado deve ser excluído permanentemente, ou seja removido do sistema de arquivos. Essa pergunta se refere apenas a um cópia de trabalho gerada a partir da camada externa que foi gerada inicialmente no diretório do projeto. Os arquivos originais, que foram utilizados para a definição não serão excluídos.
Próximos Passos
Depois do cálculo de atributos, segue uma cadeia de processos típica com a classificação temática através do painel ILMSImage Classificação.