ILMSimage 2.4 Cálculo de atributos
m |
|||
| (10 edições intermédias de um utilizador não apresentadas) | |||
| Linha 2: | Linha 2: | ||
''[[Tutorial_2.4_ILMSImage | Visão geral]]'' | ''[[Tutorial_2.4_ILMSImage | Visão geral]]'' | ||
• ''[[ILMSimage_2.4_Configurações_do_projeto | Configurações do projeto]]'' | • ''[[ILMSimage_2.4_Configurações_do_projeto | Configurações do projeto]]'' | ||
| − | • ''[[ILMSimage_2. | + | • ''[[ILMSimage_2.4_Criação_de_células | Criação de células]]'' |
• ''[[ILMSimage_2.4_Cálculo_de_atributos | Cálculo de atributos]]'' | • ''[[ILMSimage_2.4_Cálculo_de_atributos | Cálculo de atributos]]'' | ||
• ''[[IMLSimage_2.4_Classificação | Classificação]]'' | • ''[[IMLSimage_2.4_Classificação | Classificação]]'' | ||
• ''[[ILMSimage_2.4_Diversos | Diversos]]'' | • ''[[ILMSimage_2.4_Diversos | Diversos]]'' | ||
----- | ----- | ||
| + | [[en:ILMSimage_2.4_Feature_Calculation]] | ||
| + | ==[[File:ilms_img_attributes_icon.png|50px|<span title=""></span>]] Cálculo de atributos == | ||
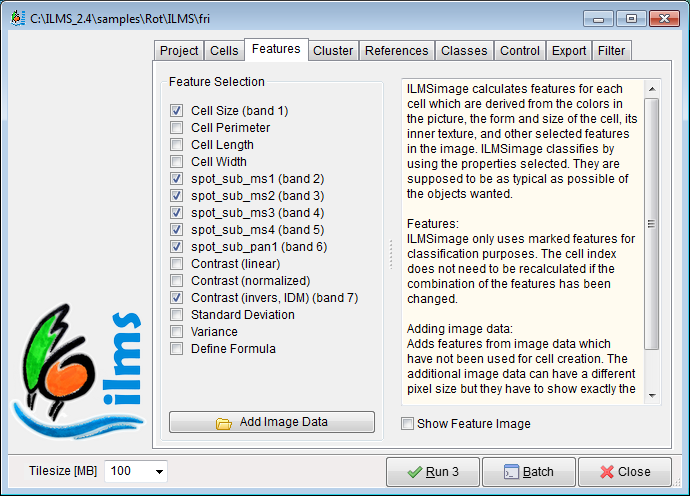
| + | A ''Seleção de recursos ILMSImage'' atribui características ou atributos (features) para cada célula no índice. Todo o trabalho de classificação depende de células e de características celulares. Os recursos podem ser calculados diretamente a partir dos dados de imagem fornecidos ou eles podem ser adicioados a partir de camadas raster externas. | ||
| + | [[File:ILMSimage_features.png]] | ||
| − | == | + | ==Parâmetros== |
| − | |||
| − | + | A ''Feature Selection'' (Seleção de Recursos) mostra uma lista completa de todos os recursos que o ILMSimage pode derivar diretamente das camadas de imagem carregadas. Para selecionar as funções para o processamento adicional, elas devem ser marcadas clicando-se nas caixas de seleção. Os quatro primeiros itens correspondem à forma das células, os seguintes disponibilizam todas as bandas de cor nas camadas de imagem carregadas e os últimos cinco itens compreendem características de textura e de desvio. O item de menu ''Add Image Data'' (Adicionar Dados de Imagem) abre novas bandas de imagens para selecionar os recursos calculados em outros lugares. | |
| − | + | ||
| − | + | ||
| − | + | *O '''Cell Size''' (Tamanho da célula) seleciona a área coberta pela célula como recurso. Todos os atributos de células são medidos em pixels. | |
| − | + | *O '''Cell Perimeter''' (Perímetro celular) seleciona o perímetro da célula medido nas fronteiras de um pixel. Em casos normais o ''tamanho da célula'' e o ''Perímetro celular'' são altamente correlacionados. | |
| − | + | *O '''Cell length''' (Comprimento celular) seleciona a medida da diagonal do menor retângulo que envolve completamente o célula | |
| − | + | *O '''Cell Width''' (Largura celular) seleciona a largura do maior quadrado cabível na célula | |
| − | + | *O '''Input Band''' (Banda de entrada) seleciona a densidade de cor média de todos os valores de pixel dentro de uma célula | |
| − | + | *O '''Contrast (linear)''' seleciona o cálculo de contraste padrão a partir da camada com a máxima resolução espacial. Se todas as camadas mostrarem a mesma resolução, ou se mais que uma camada tiverm uma resolução mais alta do que outros, o contraste é calculado usando-se o primeiro componente principal. | |
| − | + | *O '''Contrast (normalized)''' seleciona o cálculo de contraste utilizando o padrão de modulação das densidades de pixels. | |
| − | + | *O '''Contrast (invers, IDM)''' seleciona o Momento de Diferença Inversa ([http://ieeexplore.ieee.org/xpls/abs_all.jsp?tp=&isnumber=4309300&arnumber=4309314&tag=1 Haralick et al. (1973), S. 619]) para o cálculo de contrastes. | |
| − | *'''Cell Size''' | + | *A '''Standard Deviation''' (Desvio padrão) seleciona o desvio padrão de todos os pixels dentro da célula como atributo. |
| − | *'''Cell Perimeter''' | + | *A '''Variance''' seleciona a variância ou seja, o quadrado do desvio padrão como atributo. |
| − | *'''Cell | + | *O '''Define Formula''' está desativado. |
| − | *'''Cell Width''' | + | |
| − | *'''Input Band''' | + | |
| − | *'''Contrast (linear)''' | + | |
| − | *'''Contrast (normalized)''' | + | |
| − | *'''Contrast (invers, IDM)''' | + | |
| − | *'''Standard Deviation''' | + | |
| − | *'''Variance''' | + | |
| − | *'''Define Formula''' | + | |
---- | ---- | ||
| − | '''[Add Image Data]''' | + | O '''[Add Image Data]''' (Adicionar dados de imagem] abre a caixa de diálogo para a seleção de arquivo de sistema e adiciona uma ou mais camadas raster novas à lista da seleção. De acordo com a seleção de imagens no início das [[ILMSimage_2.4_Configurações_do_projeto | Configurações do projeto]], o ILMSimage armazena uma cópia dos dados selecionados na pasta do projeto e converte-os para um formato adequado. |
---- | ---- | ||
| − | '''Show Feature Image''' | + | O '''Show Feature Image''' (Mostrar Imagem do atributo) cria uma camada de imagem multibanda que representa todas as características das células selecionadas e apresenta-os na tela do QuantumGIS. A ''imagem do atributo'' vai normalizar os atributos selecionados da mesma forma que as rotinas de classificação farão, para que a informação transferida para o processo de classificação possa ser visualizada. Usuários não familiarizados com abordagens orientadas em objetos devem aproveitar a oportunidade para desevolverem uma noção sobre os differentes números numa tabela de atributos |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| + | O painel ''Control'' fornece a opção ''Show Fearures Table'' (Exibir tabela de atributos) para controlar o resultado do cálculo da função como uma tabela formatada. Se o índice celular for transformatado para um shapefile (arquivo de forma) com a opção ''Add Features to Cell Index Table'' (Adicionar atributos à tabela de índice celular) a mesma tabela estará disponível como tabela de atributos do shapefile do índice celular. | ||
| + | Os atributos podem ser recalculados sem se alterar o índice celular, mas todas as etapas subseqüentes devem ser repetidas com os valores de novos atributos. | ||
| − | == | + | ==Usando camadas adicionais de dados== |
| − | + | Além dos atributos padrão mencionados, as camadas externas de dados podem ser integradas no processo de cálculo de atributos. Para a integração, basicamente qualquer conjunto de dados disponíveis na forma de raster será adequado. Na integração dentro de cada camada e para cada célula, um valor médio será calculado e, no fim, adicionado à tabela de atributos. Isso significa que as características das camadas de dados adicionais são tratadas como equivalente às camadas raster selecionadas no [[ILMSimage_2.4_Configurações_do_projeto | início do projeto]], com a única diferença de que as camadas adicionais de dados não têm qualquer influência sobre a criação de células. Esta função é, portanto, adequada para a integração de níveis adicionais multiespectrais (ou combinações destes), que são necessários como atributos para análise adicional, mas não para a derivação das áreas de análise, isto é, as células para o processo de trabalho. Um exemplo simples é o ''[http://earthobservatory.nasa.gov/Features/MeasuringVegetation/measuring_vegetation_2.php Normalized Vegetation Difference Index, NDVI]'' que é uma medida para a atividade da vegetação. Ela pode ser calculada usando-se dois canais que se encontram no campo visual e no campo perto do infravermelho do espectro electromagnético. Como camada de dados adicional, uma imagem de NDVI baseada em pixels está disponível. A imagem seguinte mostra as áreas de alta atividade da vegetação em tons brilhantes e áreas de baixa atividade em tons de cinza escuros. | |
| Linha 62: | Linha 55: | ||
| − | + | Este conjunto de dados pode agora ser definido como uma camada de dados externa - para este efeito, ele não tem que ser carregado no QuantumGIS; basta saber o caminho correspondente. | |
| + | == Finalizando-se o Cálculo de Atributos == | ||
| + | Depois de clicar no botão [Run] os atributos selecionados são calculados e uma pequena janela com uma barra de progresso aparecerá e informará em uma mensagem sobre a operação em curso. Todas as mensagens de execução atual estarão listadas no painel de [[ILMSimage_2.4_Diversos | Controle]] e armazenadas em um arquivo de protocolo usando o nome de projeto para utilização posterior. | ||
| − | + | Se a opção ''Show Feature Image'' estiver ativa, a camada de atributos será carregada para a tela do QuantumGIS. Todos os recursos podem ser acessados numa visualização da tabela do painel de [[ILMSimage_2.4_Diversos | Controle]]. Se o cálculo de atributos característica tiver sido concluído com sucesso, o ILMSimage exibirá o painel de [[IMLSimage_2.4_Classificação | Cluster]]. | |
| − | |||
| − | |||
----- | ----- | ||
''[[Tutorial_2.4_ILMSImage | Visão geral]]'' | ''[[Tutorial_2.4_ILMSImage | Visão geral]]'' | ||
• ''[[ILMSimage_2.4_Configurações_do_projeto | Configurações do projeto]]'' | • ''[[ILMSimage_2.4_Configurações_do_projeto | Configurações do projeto]]'' | ||
| − | • ''[[ILMSimage_2. | + | • ''[[ILMSimage_2.4_Criação_de_células | Criação de células]]'' |
• ''[[ILMSimage_2.4_Cálculo_de_atributos | Cálculo de atributos]]'' | • ''[[ILMSimage_2.4_Cálculo_de_atributos | Cálculo de atributos]]'' | ||
• ''[[IMLSimage_2.4_Classificação | Classificação]]'' | • ''[[IMLSimage_2.4_Classificação | Classificação]]'' | ||
• ''[[ILMSimage_2.4_Diversos | Diversos]]'' | • ''[[ILMSimage_2.4_Diversos | Diversos]]'' | ||
----- | ----- | ||
Edição actual desde as 13h15min de 3 de Outubro de 2012
Visão geral • Configurações do projeto • Criação de células • Cálculo de atributos • Classificação • Diversos
Índice |
 Cálculo de atributos
Cálculo de atributos
A Seleção de recursos ILMSImage atribui características ou atributos (features) para cada célula no índice. Todo o trabalho de classificação depende de células e de características celulares. Os recursos podem ser calculados diretamente a partir dos dados de imagem fornecidos ou eles podem ser adicioados a partir de camadas raster externas.
Parâmetros
A Feature Selection (Seleção de Recursos) mostra uma lista completa de todos os recursos que o ILMSimage pode derivar diretamente das camadas de imagem carregadas. Para selecionar as funções para o processamento adicional, elas devem ser marcadas clicando-se nas caixas de seleção. Os quatro primeiros itens correspondem à forma das células, os seguintes disponibilizam todas as bandas de cor nas camadas de imagem carregadas e os últimos cinco itens compreendem características de textura e de desvio. O item de menu Add Image Data (Adicionar Dados de Imagem) abre novas bandas de imagens para selecionar os recursos calculados em outros lugares.
- O Cell Size (Tamanho da célula) seleciona a área coberta pela célula como recurso. Todos os atributos de células são medidos em pixels.
- O Cell Perimeter (Perímetro celular) seleciona o perímetro da célula medido nas fronteiras de um pixel. Em casos normais o tamanho da célula e o Perímetro celular são altamente correlacionados.
- O Cell length (Comprimento celular) seleciona a medida da diagonal do menor retângulo que envolve completamente o célula
- O Cell Width (Largura celular) seleciona a largura do maior quadrado cabível na célula
- O Input Band (Banda de entrada) seleciona a densidade de cor média de todos os valores de pixel dentro de uma célula
- O Contrast (linear) seleciona o cálculo de contraste padrão a partir da camada com a máxima resolução espacial. Se todas as camadas mostrarem a mesma resolução, ou se mais que uma camada tiverm uma resolução mais alta do que outros, o contraste é calculado usando-se o primeiro componente principal.
- O Contrast (normalized) seleciona o cálculo de contraste utilizando o padrão de modulação das densidades de pixels.
- O Contrast (invers, IDM) seleciona o Momento de Diferença Inversa (Haralick et al. (1973), S. 619) para o cálculo de contrastes.
- A Standard Deviation (Desvio padrão) seleciona o desvio padrão de todos os pixels dentro da célula como atributo.
- A Variance seleciona a variância ou seja, o quadrado do desvio padrão como atributo.
- O Define Formula está desativado.
O [Add Image Data] (Adicionar dados de imagem] abre a caixa de diálogo para a seleção de arquivo de sistema e adiciona uma ou mais camadas raster novas à lista da seleção. De acordo com a seleção de imagens no início das Configurações do projeto, o ILMSimage armazena uma cópia dos dados selecionados na pasta do projeto e converte-os para um formato adequado.
O Show Feature Image (Mostrar Imagem do atributo) cria uma camada de imagem multibanda que representa todas as características das células selecionadas e apresenta-os na tela do QuantumGIS. A imagem do atributo vai normalizar os atributos selecionados da mesma forma que as rotinas de classificação farão, para que a informação transferida para o processo de classificação possa ser visualizada. Usuários não familiarizados com abordagens orientadas em objetos devem aproveitar a oportunidade para desevolverem uma noção sobre os differentes números numa tabela de atributos
O painel Control fornece a opção Show Fearures Table (Exibir tabela de atributos) para controlar o resultado do cálculo da função como uma tabela formatada. Se o índice celular for transformatado para um shapefile (arquivo de forma) com a opção Add Features to Cell Index Table (Adicionar atributos à tabela de índice celular) a mesma tabela estará disponível como tabela de atributos do shapefile do índice celular.
Os atributos podem ser recalculados sem se alterar o índice celular, mas todas as etapas subseqüentes devem ser repetidas com os valores de novos atributos.
Usando camadas adicionais de dados
Além dos atributos padrão mencionados, as camadas externas de dados podem ser integradas no processo de cálculo de atributos. Para a integração, basicamente qualquer conjunto de dados disponíveis na forma de raster será adequado. Na integração dentro de cada camada e para cada célula, um valor médio será calculado e, no fim, adicionado à tabela de atributos. Isso significa que as características das camadas de dados adicionais são tratadas como equivalente às camadas raster selecionadas no início do projeto, com a única diferença de que as camadas adicionais de dados não têm qualquer influência sobre a criação de células. Esta função é, portanto, adequada para a integração de níveis adicionais multiespectrais (ou combinações destes), que são necessários como atributos para análise adicional, mas não para a derivação das áreas de análise, isto é, as células para o processo de trabalho. Um exemplo simples é o Normalized Vegetation Difference Index, NDVI que é uma medida para a atividade da vegetação. Ela pode ser calculada usando-se dois canais que se encontram no campo visual e no campo perto do infravermelho do espectro electromagnético. Como camada de dados adicional, uma imagem de NDVI baseada em pixels está disponível. A imagem seguinte mostra as áreas de alta atividade da vegetação em tons brilhantes e áreas de baixa atividade em tons de cinza escuros.
Este conjunto de dados pode agora ser definido como uma camada de dados externa - para este efeito, ele não tem que ser carregado no QuantumGIS; basta saber o caminho correspondente.
Finalizando-se o Cálculo de Atributos
Depois de clicar no botão [Run] os atributos selecionados são calculados e uma pequena janela com uma barra de progresso aparecerá e informará em uma mensagem sobre a operação em curso. Todas as mensagens de execução atual estarão listadas no painel de Controle e armazenadas em um arquivo de protocolo usando o nome de projeto para utilização posterior.
Se a opção Show Feature Image estiver ativa, a camada de atributos será carregada para a tela do QuantumGIS. Todos os recursos podem ser acessados numa visualização da tabela do painel de Controle. Se o cálculo de atributos característica tiver sido concluído com sucesso, o ILMSimage exibirá o painel de Cluster.
Visão geral • Configurações do projeto • Criação de células • Cálculo de atributos • Classificação • Diversos