ILMSimage 2.4 Criação de células
(Criou nova página com '----- '' Visão geral'' • '' Configurações do projeto'' • ''[[ILMSimage_2.4_Criação_de_c...') |
|||
| Linha 8: | Linha 8: | ||
----- | ----- | ||
| + | ==[[File: ilms_img_cells_icon.png|50px|<span title=""></span>]] Criação de células== | ||
| − | + | O ''cell index'' (índice celular) resume áreas com características de pixel semelhantes como "células" na imagem. A análise de imagem com o ILMS depende completamente das células. O painel de entrada ''cells'' é utilizado para controlar o tamanho da célula e a distribuição de células com três parâmetros de entrada. As ''External Borders'' (Fronteiras Externas) são influenciadas por células recém-criadas. | |
| − | + | Página principal do [[ILMSImage_2.4_Tutorial | ILMSImage 2.4. Tutorial]]. | |
| − | |||
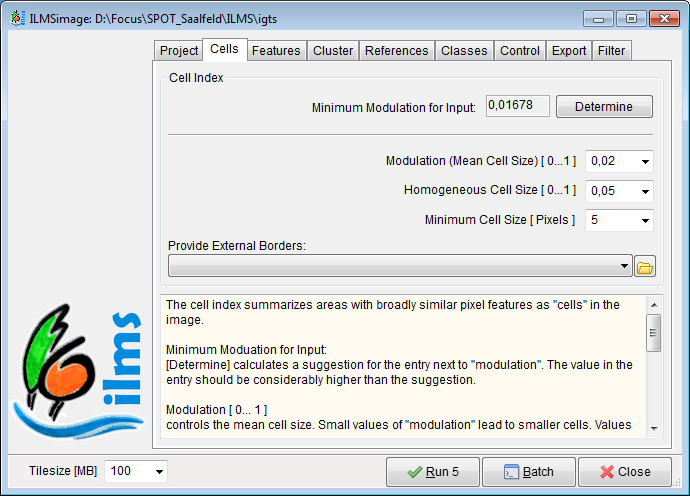
| + | [[File:ILMSimage_CellCreation.png|Painel ILMSimage Criação de células]] | ||
| − | |||
| + | == Parâmetros == | ||
| − | + | *'''Minimum Modulation for input''' (Modulação mínima de entrada). É difícil acertar um valor adequado para a entrada na ''Modulation (tamanho médio das células)'', de modo que o botão ''Determine'' inicia uma rotina que calcula a média da modulação das imagens selecionadas. O resultado é exibido na caixa à esquerda do botão. A entrada na ''Modulation (tamanho médio das células) deve ser consideravelmente maior do que o resultado da ''modulação mínima de entrada'' . | |
| + | *'''Modulation (Mean Cell Size)''' é o parâmetro de entrada mais sigificante. A entrada determina o tamanho de todas as superfícies resultantes homogéneas referidas como "células", resultantes do processo de criação de células. Os menores valores de ''modulação'' criam células menores. Os valores de entrada 0-1 estão definidos. | ||
| + | *'''Tamanho da Célula Homogéneo''' determina a distribuição dos tamanhos de células diferentes, após o processo de criação de células. Pequenos valores (0,0 - 0,1) conduzem a uma distribuição de tamanho "natural", com áreas muito diferentes, cobertas por uma única célula. Este é o caso normal, mas pode ser útil manter os tamanhos de célula iguais, a fim de executar-se as tarefas de classificação incomuns. | ||
| + | *O '''Minimum Cell Size''' (Tamanho mínimo de célula) assegura que todas as células consistam pelo menos da quantidade de pixles da entrada. Se as células são menores do que a entrada, o ILMSimage distribui os pixels igualmente entre as células vizinhas. Este processo é independente de dados de imagem. | ||
| + | *O '''Provide External Borders''' (Fornecer Fronteiras externas) transfere todas as bordas de uma dada forma para as células. O ILMSimage pode adicionar bordas adicionais dentro das células predestinadas. A entrada das ''Fronteiras Externas'' é opcional. | ||
| − | + | O ILMSImage utiliza um conceito de tile (telha)CONFERIREUACHOQUENAOSEDIZTELHA para processar grandes quantidades de camadas de dados eficientemente. O tamanho máximo das telhas aplicadas pode ser limitado por uma entrada em '''TileSize [MB]''', no canto inferior esquerdo do menu ILMSimage. Em condições normais, o ILMSimage usa até 10 telhas simultâneamente, dessa forma para cada 1000 MB de memória de trablho livre, uma entrada de "100 MB" é recomendada. | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | |||
| + | == Finalizando a criação de células == | ||
| − | + | Depois de clicar no botão [Run] as operações selecionadas são realizadas e uma pequena janela com uma barra de progresso aparecerá e fornecerá informações sobre o estado da geração de células. | |
| − | + | ||
| − | + | ||
[[File: ILMSimage_ProgressBar.png]] | [[File: ILMSimage_ProgressBar.png]] | ||
| − | + | Isto pode levar algum tempo, de acordo com o tamanho da área de teste e os parâmetros selecionados. | |
| − | + | Após o processo de criação de células terminar o QuantumGIS mostra as bordas das células resultantes com uma camada raster SIG semitransparente. Camadas raster podem ser visualizadas consideravelmente mais rapidamente do que as camadas de vetor com bastantes polígonos. Se a criação de células terminar com sucesso, o ILMSimage mostrará o painel [[ILMSimage_2.4_Cálculo_de_atributos | cálculo de atributos]]. | |
| − | + | Se as bordas de células tiverem que ser representadas por formas, o painel do ILMSimage '''[Export]''' fornece ferramentas para converter o índice de células rasterizadas em uma camada de forma e mostrá-lo na tela QuantumGIS. É possível suavizar os polígonos gerados adaptativamente, o que pode ser feito ativando a caixa de seleção ''Smooth arcs [grade]'' (arcos suaves [grau]) e ''Optimize Vertex Density'' (otimizar densidade do Vertex). O grau de suavizamento pode ser determinado na caixa de entrada, onde os polígonos são mais fortemente suavizados com grau crescente. | |
| − | == | + | ==Antecedentes == |
| − | + | A derivação de áreas de imagem estatisticamente homogêneas é um '"processo-chave em uma análise de imagem baseada em objeto'''. Ela se baseia no pressuposto de que o material de dados de modernos sensores de sensoriamento remoto, o qual está cada vez mais complexo, devido ao aumento da densidade de dados, não pode ser analisado de forma adequada, porque os pixels da análise de imagem comum são utilizados como referência. Como uma alternativa, a análise de imagem baseada em objetos oferece a criação de novas áreas como base para a análise subsequente. Este processo é geralmente chamado de '"segmentação''' e, no contexto do ILMSImage como '''criação de células'''. Ele sintetiza pixels vizinhos, com base em semelhanças de estatística para áreas (células) que são tão homogénea quanto possível. Este processo não representa apenas uma redução da complexidade dos dados de entrada abrangentes, mas também oferece uma nova perspectiva sobre esses dados, fornecendo novos recursos (que raramente são usados em análise de imagem) para posterior análise. Uma dessas características é, por exemplo, sob a forma de uma célula ou seu contexto espacial, que contém, entre outras coisas, a função das células vizinhas. Atributos para forma e contexto são percebidos como características subordinados em análise baseada em pixels de dados de sensoriamento remoto, devido à uniformidade da área, principalmente do pixel retangular. | |
| − | + | ||
| − | + | Para o processo de segmentação, isto é, a derivação de tais áreas de referência a partir de imagens diferentes, sugestões e métodos são dados na literatura correspondente. Alguns deles encontram-se resumidos em [http://www.sciencedirect.com/science/article/pii/S0734189X85901537 Haralick & Shapiro (1985)], [http://www.sciencedirect.com/science/article/pii/003132039390135J Pal & Pal (1993)] e [http://www.springerlink.com/content/311jw5m3a1fr6a4w/ Freixenet et al. (2002)]. Para uma compreensão básica do ILMSImage basta saber que o processo de criação de células utilizado no software baseia-se numa combinação dos métodos Region Growing and Watershed Analysis (crescimento de região e Análise de Watershed) . Ela foi desenvolvida com o objetivo de minimizar os graus de liberdade, disponíveis para o usuário para a adaptação. Para uma aplicação exata do método, opções mais detalhadas estão disponíveis. O resultado da criação de células utilizando o ILMSImage é uma abstracção do primeiro conteúdo de imagem original, todas as outras análises são realizadas utilizando esta imagem de célula. | |
| − | == | + | == Modulação == |
| − | + | O termo '''Modulation''' deriva do contraste máximo entre pixels vizinhos nos dados de imagem ([http://books.google.com/books?id=KQXNaDH0X-IC&lpg=PA147&vq=modulation&dq=MS%20Remote%20Sensing&hl=de&pg=PA146#v=onepage&q&f=true Schowengerdt, 2007]). O parâmetro de modulação controla o tamanho médio das células emergentes. A seguinte relação tem de ser considerada: com uma crescente modulação o tamanho médio das células aumenta, ao passo que o seu número diminui, consequentemente se a modulação diminui o tamanho médio das células também diminui, mas o seu número aumenta. | |
| − | + | A gama de valores do parâmetro situa-se entre 0 e 1, note-se que os valores extremos não conduzem a resultados significantes sob quaisquer circunstâncias. O valor 0,03 é um valor pré-definido de modulação, que tem provado ser um bom ponto de partida para a criação de células de uma combinação de dados pancromáticos de maior resolução espacial e dados multi-espectrais de menor resolução espacial (como eles são utilizados no exemplo atual). Essa configuração padrão pode ser modificada pelo usuário sobre a criação de parâmetros de células, de modo que ele corresponda ao tamanho da área que é tipicamente necessária para a análise. O resultado de uma nova criação de célula é um conjunto de dados raster, que tem o tamanho do pixel da maioria dos dados de entrada de alta resolução, que podem ser encontrados no diretório do projeto e termina em <code>_index</code>. Como o ILMSImage requer acesso exclusivo para o arquivo correspondente para mais operações, ele não é automaticamente carregado na visualização do mapa. Para visualizar o resultado da criação de células, a opção de exportação como Shapefile (ver abaixo) é mais adequada. | |
----- | ----- | ||
Revisão das 12h00min de 10 de Setembro de 2012
Visão geral • Configurações do projeto • Criação de células • Cálculo de atributos • Classificação • Diversos
Índice |
 Criação de células
Criação de células
O cell index (índice celular) resume áreas com características de pixel semelhantes como "células" na imagem. A análise de imagem com o ILMS depende completamente das células. O painel de entrada cells é utilizado para controlar o tamanho da célula e a distribuição de células com três parâmetros de entrada. As External Borders (Fronteiras Externas) são influenciadas por células recém-criadas.
Página principal do ILMSImage 2.4. Tutorial.
Parâmetros
- Minimum Modulation for input (Modulação mínima de entrada). É difícil acertar um valor adequado para a entrada na Modulation (tamanho médio das células), de modo que o botão Determine inicia uma rotina que calcula a média da modulação das imagens selecionadas. O resultado é exibido na caixa à esquerda do botão. A entrada na Modulation (tamanho médio das células) deve ser consideravelmente maior do que o resultado da modulação mínima de entrada .
- Modulation (Mean Cell Size) é o parâmetro de entrada mais sigificante. A entrada determina o tamanho de todas as superfícies resultantes homogéneas referidas como "células", resultantes do processo de criação de células. Os menores valores de modulação criam células menores. Os valores de entrada 0-1 estão definidos.
- Tamanho da Célula Homogéneo determina a distribuição dos tamanhos de células diferentes, após o processo de criação de células. Pequenos valores (0,0 - 0,1) conduzem a uma distribuição de tamanho "natural", com áreas muito diferentes, cobertas por uma única célula. Este é o caso normal, mas pode ser útil manter os tamanhos de célula iguais, a fim de executar-se as tarefas de classificação incomuns.
- O Minimum Cell Size (Tamanho mínimo de célula) assegura que todas as células consistam pelo menos da quantidade de pixles da entrada. Se as células são menores do que a entrada, o ILMSimage distribui os pixels igualmente entre as células vizinhas. Este processo é independente de dados de imagem.
- O Provide External Borders (Fornecer Fronteiras externas) transfere todas as bordas de uma dada forma para as células. O ILMSimage pode adicionar bordas adicionais dentro das células predestinadas. A entrada das Fronteiras Externas é opcional.
O ILMSImage utiliza um conceito de tile (telha)CONFERIREUACHOQUENAOSEDIZTELHA para processar grandes quantidades de camadas de dados eficientemente. O tamanho máximo das telhas aplicadas pode ser limitado por uma entrada em TileSize [MB], no canto inferior esquerdo do menu ILMSimage. Em condições normais, o ILMSimage usa até 10 telhas simultâneamente, dessa forma para cada 1000 MB de memória de trablho livre, uma entrada de "100 MB" é recomendada.
Finalizando a criação de células
Depois de clicar no botão [Run] as operações selecionadas são realizadas e uma pequena janela com uma barra de progresso aparecerá e fornecerá informações sobre o estado da geração de células.
Isto pode levar algum tempo, de acordo com o tamanho da área de teste e os parâmetros selecionados.
Após o processo de criação de células terminar o QuantumGIS mostra as bordas das células resultantes com uma camada raster SIG semitransparente. Camadas raster podem ser visualizadas consideravelmente mais rapidamente do que as camadas de vetor com bastantes polígonos. Se a criação de células terminar com sucesso, o ILMSimage mostrará o painel cálculo de atributos.
Se as bordas de células tiverem que ser representadas por formas, o painel do ILMSimage [Export] fornece ferramentas para converter o índice de células rasterizadas em uma camada de forma e mostrá-lo na tela QuantumGIS. É possível suavizar os polígonos gerados adaptativamente, o que pode ser feito ativando a caixa de seleção Smooth arcs [grade] (arcos suaves [grau]) e Optimize Vertex Density (otimizar densidade do Vertex). O grau de suavizamento pode ser determinado na caixa de entrada, onde os polígonos são mais fortemente suavizados com grau crescente.
Antecedentes
A derivação de áreas de imagem estatisticamente homogêneas é um '"processo-chave em uma análise de imagem baseada em objeto. Ela se baseia no pressuposto de que o material de dados de modernos sensores de sensoriamento remoto, o qual está cada vez mais complexo, devido ao aumento da densidade de dados, não pode ser analisado de forma adequada, porque os pixels da análise de imagem comum são utilizados como referência. Como uma alternativa, a análise de imagem baseada em objetos oferece a criação de novas áreas como base para a análise subsequente. Este processo é geralmente chamado de '"segmentação e, no contexto do ILMSImage como criação de células. Ele sintetiza pixels vizinhos, com base em semelhanças de estatística para áreas (células) que são tão homogénea quanto possível. Este processo não representa apenas uma redução da complexidade dos dados de entrada abrangentes, mas também oferece uma nova perspectiva sobre esses dados, fornecendo novos recursos (que raramente são usados em análise de imagem) para posterior análise. Uma dessas características é, por exemplo, sob a forma de uma célula ou seu contexto espacial, que contém, entre outras coisas, a função das células vizinhas. Atributos para forma e contexto são percebidos como características subordinados em análise baseada em pixels de dados de sensoriamento remoto, devido à uniformidade da área, principalmente do pixel retangular. Para o processo de segmentação, isto é, a derivação de tais áreas de referência a partir de imagens diferentes, sugestões e métodos são dados na literatura correspondente. Alguns deles encontram-se resumidos em Haralick & Shapiro (1985), Pal & Pal (1993) e Freixenet et al. (2002). Para uma compreensão básica do ILMSImage basta saber que o processo de criação de células utilizado no software baseia-se numa combinação dos métodos Region Growing and Watershed Analysis (crescimento de região e Análise de Watershed) . Ela foi desenvolvida com o objetivo de minimizar os graus de liberdade, disponíveis para o usuário para a adaptação. Para uma aplicação exata do método, opções mais detalhadas estão disponíveis. O resultado da criação de células utilizando o ILMSImage é uma abstracção do primeiro conteúdo de imagem original, todas as outras análises são realizadas utilizando esta imagem de célula.
Modulação
O termo Modulation deriva do contraste máximo entre pixels vizinhos nos dados de imagem (Schowengerdt, 2007). O parâmetro de modulação controla o tamanho médio das células emergentes. A seguinte relação tem de ser considerada: com uma crescente modulação o tamanho médio das células aumenta, ao passo que o seu número diminui, consequentemente se a modulação diminui o tamanho médio das células também diminui, mas o seu número aumenta.
A gama de valores do parâmetro situa-se entre 0 e 1, note-se que os valores extremos não conduzem a resultados significantes sob quaisquer circunstâncias. O valor 0,03 é um valor pré-definido de modulação, que tem provado ser um bom ponto de partida para a criação de células de uma combinação de dados pancromáticos de maior resolução espacial e dados multi-espectrais de menor resolução espacial (como eles são utilizados no exemplo atual). Essa configuração padrão pode ser modificada pelo usuário sobre a criação de parâmetros de células, de modo que ele corresponda ao tamanho da área que é tipicamente necessária para a análise. O resultado de uma nova criação de célula é um conjunto de dados raster, que tem o tamanho do pixel da maioria dos dados de entrada de alta resolução, que podem ser encontrados no diretório do projeto e termina em _index. Como o ILMSImage requer acesso exclusivo para o arquivo correspondente para mais operações, ele não é automaticamente carregado na visualização do mapa. Para visualizar o resultado da criação de células, a opção de exportação como Shapefile (ver abaixo) é mais adequada.
Visão geral • Configurações do projeto • Criação de células • Cálculo de atributos • Classificação • Diversos